Im Rahmen unserer Behandlung der Molekularbiologie im Unterricht besuchten wir, der Biologiegrundkurs (Q1) von Frau Ackermann, am 27.11.19, das Lernlabor der TU-Darmstadt. Unter anderem ist es der Anspruch des Labors Schülerinnen und Schülern, durch eigenständiges Experimentieren, neue Erkenntnisse in der modernen Biologie gewinnen zu lassen.
Indessen wurden uns die wichtigsten molekularbiologischen Methoden präsentiert. Diese wurden durch Herrn Dr. Guido Klees vorgestellt, worauf wir auch gleich mit unserem eigenen Erproben begannen. Mit der Unterstützung von Herrn Dr. Klees, zwei hilfsbereiten Studenteninnen der TU und unserer Lehrerin sollten wir nun ein Plasmid des Bakteriums Escherichia coli analysieren.
Einfacher gesagt als getan. Das Plasmid, welches mit dem GFP-Gen (green fluoreszent protein) versehen wurde, galt es zu isolieren, was durch das Aufschließen der E. coli-Zellen erreicht wurde.
Durch die Methode der Polymerase-Kettenreaktion (PCR), wurde nun das isolierte Plasmid mitsamt dem fluoreszierenden Gen vervielfältigt. Dies wurde durch die Zugabe von DNA-Polymerase und Primern durch uns synthetisch herbeigeführt.
Nachdem all die vorbereitenden Schritte getroffen waren, konnten wir nun an die Restriktionsanalyse herangehen. Das Plasmid wurde hier einmal mit dem Restriktionsenzym EcoRI an einer definierten Sequenz geschnitten, wodurch die ringförmige Ursprungsform durch eine lineare ersetzt wird, welche mitunter für die Gelelektrophorese erforderlich ist. Als nächstes wurde das Enzym HindIII zur Überprüfung der Orientierung des GFP-Gens eingesetzt.
Danach ging es an die Vorbereitung der Gelelektrophorese, wofür wir zunächst einmal eine Lösung mit Agarose-Pulver herstellten. Bevor wir uns nach der einstündigen Mittagspause wieder zurück ins Labor begaben, sollten wir, mithilfe einer digitalen Plasmidanalyse, in der die Größe der DNA-Fragmente angegeben war, Vorhersagen über die Orientierung der DNA-Banden treffen.
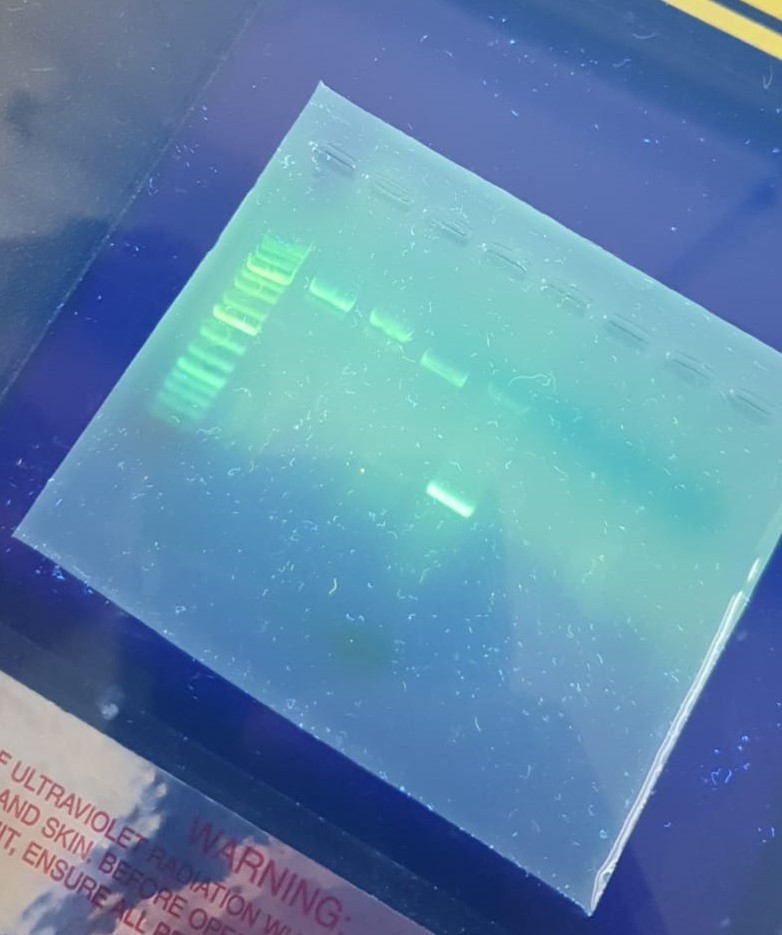
Danach ging es wieder daran, das ganze praktisch im Labor zu bestätigen. Die hergestellte Lösung bildete mittlerweile ein Gel, sodass wir mit den Vorbereitungen zur Elektrophorese fortfahren konnten. Hierfür pipettierten wir verschiedene Proben in die Geltaschen (Bild 3). Diese waren einmal ein Marker zur DNA-Größenbestimmung, eine Plasmidlösung ohne Restriktionsenzyme zur Kontrolle, die, mit EcoRI und HindIII geschnittene Probe sowie das in der PCR vervielfältigte GFP-Gen. Nun konnten wir die Elektrophorese, bei der, wie von Herrn Dr. Klees erklärt, die negativ geladene DNA sich in einem elektrischen Feld in Richtung der Anode, je nach Größe weiter oder kürzer bewegt, starten. An einer Geldokumentationsstation (Bild 4) ließen sich nun, durch die Ergebnisse, unsere Vorhersagen bestätigen.
Rückblickend lässt sich sagen, dass es eine sehr lehrreiche Exkursion war. Zum einen wurden wir an das eigenständige Experimentieren in einem Labor herangeführt und zum anderen lernten wir moderne Forschungsmethoden kennen. Des Weiteren dienten die, an diesem Tag gewonnenen Erkenntnisse, als Ergänzung unserer Themen aus dem Unterricht.
Verf. von Noah Baumann/Franziska Monath